Fiocruz: BA.2, variante mais transmissível da ômicron, já chegou ao Brasil; no Reino Unido e Dinamarca, causou explosão de casos
Por Conceição Lemes
A Rede Genômica Fiocruz divulgou nesta sexta-feira, 04-02, novos dados sobre a situação das linhagens e variantes do vírus da covid-19 no Brasil.
O levantamento demonstra:
1. O quão rápido a ômicron dominou o Brasil.
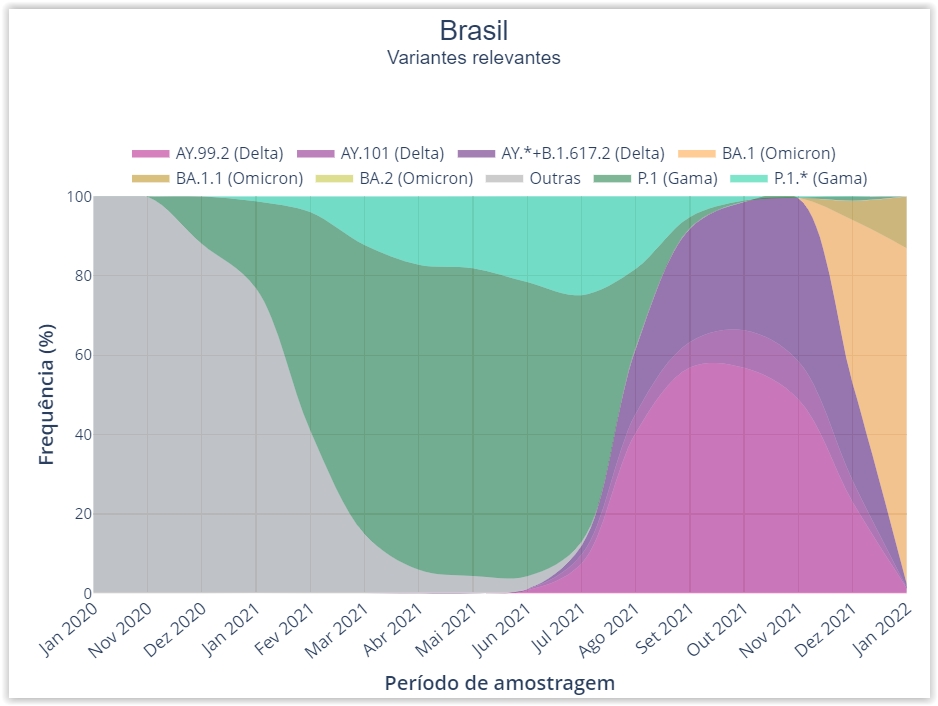
Em dezembro de 2021, representava 39,4% das infecções. Em janeiro, esse índice já tinha chegado a 95,9%.
2. A BA.2, nova variante da ômicron, já chegou ao Brasil.
A Fiocruz detectou um caso entre as amostras coletadas entre 14 e 27 de janeiro.
A BA.2 é 33% mais transmissível que a BA.1, a linhagem original da ômicron.
Além disso, a BA.2 tem maior capacidade de infectar pessoas já vacinadas.
A BA.2 está associada a uma explosão de casos na Dinamarca e no Reino Unido.
Nos EUA, foram detectados 127 casos até o momento.
Abaixo, a íntegra da nota da Agência Fiocruz de Notícias sobre os novos dados da Rede Genômica.
Variante Ômicron representa mais de 95% dos genomas sequenciados no país
Por Ricardo Valverde, Agência Fiocruz de Notícias
A Rede Genômica Fiocruz divulgou novos dados sobre a situação das linhagens e variantes do vírus SARS-CoV-2 no Brasil, tendo em vista os resultados da vigilância genômica produzidos pela Rede e por outras iniciativas.
De acordo com a publicação, se em dezembro a variante Ômicron representou 39,4% dos genomas sequenciados, em janeiro de 2022 esse índice chegou a 95,9%, sendo encontrada em todas as regiões do país.
O Relatório da Rede mostra que a Ômicron domina completamente o cenário epidemiológico da Covid-19 no Brasil. A publicação também destaca inovações tecnológicas desenvolvidas pela Rede Genômica Fiocruz.
Nas duas semanas (de 14 a 27 de janeiro) a que se referem os dados divulgados pela Rede, o Laboratório de Vírus Respiratórios e Sarampo do Instituto Oswaldo Cruz (IOC/Fiocruz) e unidades da Fundação em seis estados (Amazonas, Ceará, Pernambuco, Paraná, Bahia e Minas Gerais) produziram 3.739 genomas.
Cada uma das unidades de sequenciamento da Fiocruz (além das já citadas, há também uma no Piauí) atende uma ou mais Unidade da Federação (UF).
Os pesquisadores da Rede Genômica Fiocruz lembram que todo esse monitoramento de vigilância é possível devido a parceria e colaboração dos Laboratorios Estaduais de Saúde Pública (LACENs) e da Coordenação-Geral de Laboratórios do Ministério da Saúde. Também a plataforma GISAID de depósitos de genomas da OMS contribui muito para que este depósito ocorra em tempo real.
A amostragem utilizada pela Rede Genômica Fiocruz em apoio à rede nacional de vigilância de vírus respiratórios está baseada em dois pilares fundamentais.
Um é a amostragem representativa dos casos positivos para SARS-CoV-2 por teste RT-PCR em tempo real realizados em cada UF.
Outro são as amostras de eventos inusitados identificados pelas vigilâncias locais.
Há atualmente mais de mil linhagens definidas do vírus SARS-CoV-2, mas apenas 5 foram identificadas como variantes de preocupação, com impacto significativo na saúde pública, devido a características como maior capacidade de transmissão e infecção, maior capacidade de escape de anticorpos ou uma combinação destas.
Os primeiros genomas da Ômicron no Brasil são de amostras do fim de novembro e ao término de dezembro a variante já era a mais frequente nas regiões Sudeste, Nordeste e Sul. No momento, a Ômicron é classificada em quatro linhagens (BA.1, BA.1.1, BA.2 e BA.3).
No Brasil, até o fechamento da nova edição do Relatório da Rede Genômica Fiocruz, foram identificadas as linhagens BA.1 (2.382 genomas), BA.1.1 (226 genomas) e BA.2 (1 genoma).
ViralFlow
O Relatório informa que a Rede Genômica Fiocruz desenvolveu a plataforma ViralFlow, com o objetivo de mudar o cenário em que grupos de pesquisa trabalham de forma descentralizada, com diferentes ferramentas para obter e analisar as sequências. O trabalho baseado em ferramentas dispersas consome tempo e exige que pesquisadores invistam no treinamento para utilização de vários serviços.
Além disso, em casos em que um mesmo paciente está infectado com duas ou mais variantes, a separação e a montagem dos genomas não é possível por meio dessas ferramentas.
Desenvolvido na Fiocruz Pernambuco, o ViralFlow automatiza vários processos importantes para a vigilância genômica e oferece uma plataforma para que pesquisadores possam estudar múltiplos aspectos de amostras do SARS-CoV-2 de forma centralizada e ágil.
Helper
Outra inovação, visando aperfeiçoar o trabalho da Rede Genômica Fiocruz, é o sistema Helper, empregado de forma bem-sucedida em ensaios diagnósticos e testes de inferência para detecção de variantes.
A ferramenta proporciona maior eficiência nas conferências múltiplas de resultados e no tempo de cadastro e liberação de resultados (aproximadamente quatro horas de um analista por cada bateria de resultados gerados).
O processo de implementação é conduzido pela Fiocruz Ceará. O uso da ferramenta permitirá a descentralização em diversos laboratórios, incluindo os Lacen, e a realização de exercícios de vigilância laboratorial, particularmente ensaios de inferência para variantes do Sars-CoV-2.
Testes de inferência
No site da Rede Genômica Fiocruz foram incluídos os dados de RT-PCR de inferência para variantes realizados pelos Lacen.
Já foram realizados 3.984 testes em 16 estados (Acre, Amazonas, Amapá, Roraima, Bahia, Ceará, Pará, Rio de Janeiro, Paraíba, Santa Catarina, Rio Grande do Norte, Rondônia, Rio Grande do Sul, Sergipe, São Paulo e Tocantins), de novembro de 2021 a janeiro 2022.
Um dos ensaios para a realização desses testes, para detecção de Sars-CoV-2 e triagem de variantes, foi desenvolvido pelo Laboratório de Tecnologia Diagnóstica do Instituto de Tecnologia em Imunobiológicos (Bio-Manguinhos/Fiocruz) em parceria com o Laboratório de Vírus Respiratórios e Sarampo do IOC/Fiocruz.

Publicação de: Viomundo
